肠道微生态的研究策略是一个多维度、多层次的系统性工程,需要整合微生物学、分子生物学、基因组学、代谢组学、免疫学、临床医学等多个学科的技术与方法,其核心目标是解析肠道微生物与宿主之间的相互作用关系,揭示其在健康维持与疾病发生发展中的作用机制,并基于此开发诊断、治疗及预防新策略,以下从不同层面详细阐述其研究策略。
在样本采集与标准化处理方面,这是确保研究结果可靠性的基础,样本类型包括粪便、肠道黏膜组织、肠腔内容物等,其中粪便样本因无创、易获取且能反映肠道微生物群落整体特征,成为最常用的研究样本,粪便微生物与肠道黏膜微生物存在差异,因此在特定研究中需结合黏膜活检样本,采集过程中需严格控制变量,如采集容器、保存温度(80℃)、保存时间以及运输条件,以避免微生物死亡或群落结构改变,需详细记录受试者的基本信息(年龄、性别、饮食、生活习惯)、疾病状态、用药史(尤其是抗生素、益生菌等)等 metadata,这些信息对于后续数据分析和结果解释至关重要,标准化操作流程(SOP)的建立,包括样本前处理(如 DNA 提取方法的选择、去除抑制剂)等,是保证不同研究间结果可比性的前提。
在微生物群落结构与功能分析方面,高通量测序技术是当前的核心手段,16S rRNA 基因测序因其 V3-V4 等高变区能够较好地区分细菌属种,具有成本低、通量高的优点,广泛应用于微生物群落多样性(α多样性:反映群落丰富度和均匀度;β多样性:反映不同样本间群落结构差异)和物种组成分析,16S 测序无法在种水平精确定量,且无法检测真菌、病毒等其他微生物,宏基因组测序则通过直接提取样本总 DNA 并进行高通量测序,能够获得微生物物种组成(包括细菌、古菌、真菌、病毒等)、功能基因(如碳水化合物活性酶 CAZymes、抗生素抗性基因 ARGs、毒力因子 VF)及代谢通路信息,全面反映微生物群落的遗传潜力,近年来,宏转录组学(研究微生物的转录活性)、宏蛋白质组学(研究微生物及宿主的蛋白质表达)和代谢组学(检测微生物代谢产物如短链脂肪酸 SCFAs、色氨酸代谢物、胆汁酸等)的发展,进一步从功能层面揭示了微生物群落的动态变化及其与宿主的相互作用,短链脂肪酸不仅是微生物代谢产物,还能作为信号分子调节宿主免疫和代谢功能。
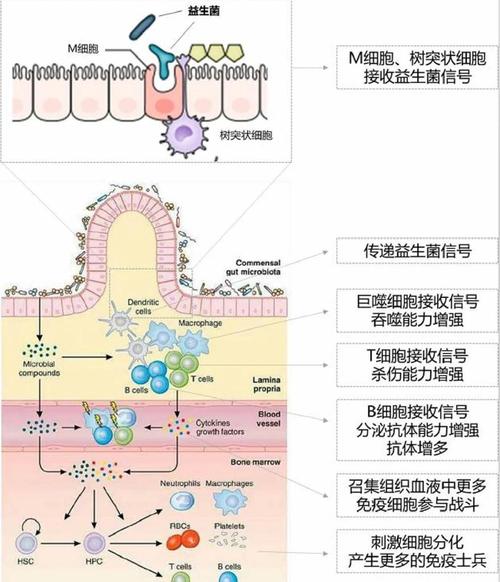
在肠道微生态与宿主互作的机制研究中,需要结合体外模型和体内模型,体外模型包括肠道类器官(organoid)、肠道上皮细胞单层培养(如 Caco-2 细胞)、厌氧发酵系统(如 SHIME 系统,模拟人体肠道环境)等,可用于研究特定微生物或代谢产物对肠道上皮屏障、免疫细胞功能的影响,体内模型主要包括无菌动物(Germ-free mice,肠道内无微生物定植)、抗生素处理动物、特定微生物定植动物(如人类来源微生物移植 HMMF)以及基因工程动物等,通过这些模型,可以阐明微生物在宿主免疫系统发育、代谢调控(如能量代谢、脂质代谢、葡萄糖稳态)、神经系统调节(肠-脑轴)等方面的具体作用机制,将肥胖患者的肠道微生物移植给无菌小鼠,可导致受体小鼠出现肥胖表型,直接证明了肠道微生物在肥胖发生中的因果作用。
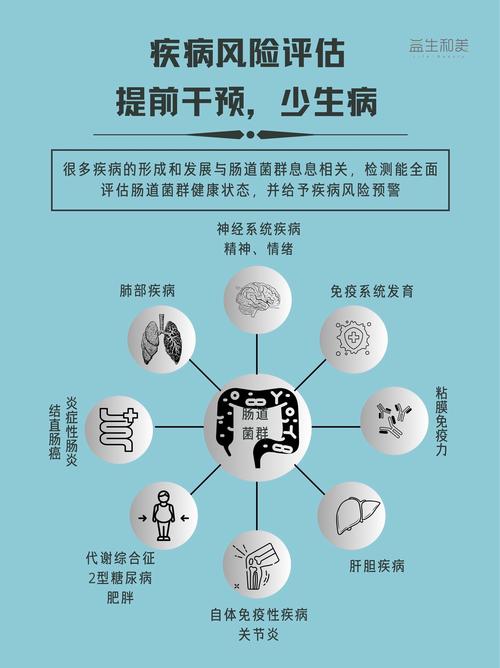
在临床转化与应用研究方面,肠道微生态研究致力于将基础发现转化为临床应用,诊断策略上,基于微生物标志物(如特定菌群的丰度变化、功能基因的异常表达)开发疾病诊断工具,如结直肠癌、炎症性肠病(IBD)、肝硬化的无创诊断方法,治疗策略上,包括粪菌移植(FMT,已用于复发性艰难梭菌感染的治疗,并在 IBD、肠易激综合征等疾病中显示出潜力)、益生菌(特定有益菌株,如双歧杆菌、乳酸杆菌)、益生元(不可被宿主消化但能促进有益菌生长的物质,如低聚果糖)、合生元(益生菌与益生元的组合)以及基于微生物代谢产物(如丁酸盐)的治疗,药物开发方面,靶向微生物或其代谢产物的小分子药物正在探索中,例如抑制有害菌的毒力因子或调节微生物代谢通路以增强疗效。
在数据整合与系统生物学分析层面,肠道微生态研究产生的多组学数据(基因组、转录组、代谢组、临床数据等)具有高维度、复杂性特点,需要运用生物信息学工具和统计学方法进行数据预处理、质量控制、差异分析、功能富集分析、网络分析(如微生物-微生物互作网络、微生物-宿主代谢物关联网络)等,机器学习和人工智能算法(如随机森林、深度学习)可用于挖掘微生物标志物组合,构建疾病预测模型,系统生物学方法则有助于从整体水平理解肠道微生态作为一个“微生物器官”与宿主形成的复杂生态系统。
以下表格总结了肠道微生态研究中常用的技术方法及其应用:
| 技术类型 | 常用方法/技术 | 主要应用领域 |
|---|---|---|
| 微生物群落分析 | 16S rRNA 基因测序 | 微生物物种组成、多样性分析(α多样性、β多样性) |
| 宏基因组测序 | 物种鉴定(细菌、真菌、病毒等)、功能基因注释、代谢通路分析 | |
| 功能活性分析 | 宏转录组测序 | 微生物基因表达谱、功能活性研究 |
| 代谢组学(LC-MS, GC-MS) | 微生物及宿主代谢产物检测(SCFAs、氨基酸、胆汁酸等) | |
| 机制研究 | 体外模型(类器官、细胞培养) | 微生物-上皮细胞互作、代谢产物功能验证 |
| 体内模型(无菌动物、基因工程动物) | 微生物在宿主生理病理中的作用机制、因果关系验证 | |
| 临床转化 | 粪菌移植(FMT) | 复发性艰难梭菌感染、IBD 等疾病治疗 |
| 益生菌/益生元/合生元 | 调节肠道微生态平衡,改善宿主健康 | |
| 数据分析 | 生物信息学(QIIME, MetaPhlAn, LEfSe) | 数据处理、物种注释、差异分析、功能富集 |
| 机器学习/系统生物学 | 生物标志物挖掘、模型构建、生态系统网络分析 |
相关问答 FAQs:
问题 1:肠道微生态研究中,16S rRNA 基因测序和宏基因组测序各有何优缺点?如何选择?
解答:16S rRNA 基因测序的主要优点是成本较低、通量高、数据分析相对简单,能够快速获取样本中细菌群落的组成和多样性信息,适用于大样本量的群体验证和初步筛选,缺点是分辨率有限,通常只能鉴定到属或科水平,无法准确区分种水平,且无法检测非细菌微生物(如真菌、病毒)和功能基因信息,宏基因组测序的优点是分辨率高,能够达到种甚至株水平,可全面分析细菌、古菌、真菌、病毒等微生物群落,并能进行功能基因注释和代谢通路分析,直接反映微生物的遗传潜力,缺点是成本较高、数据分析复杂、对 DNA 质量和量要求高,且无法区分活菌和死菌,选择时,若研究目标是初步了解微生物群落结构变化或进行大样本量筛查,16S 测序是经济高效的选择;若需深入研究物种精细组成、功能基因或涉及非细菌微生物,则应选择宏基因组测序。
问题 2:粪菌移植(FMT)在肠道微生态相关疾病治疗中面临哪些挑战?
解答:粪菌移植(FMT)虽然在一些疾病(如复发性艰难梭菌感染)中取得了显著疗效,但仍面临诸多挑战:① 供体筛选与标准化:目前尚无统一的供体筛选标准,包括健康状态、饮食、生活习惯等,供体的异质性可能导致疗效差异和潜在风险(如传播未知病原体)。② 移植方式与剂型优化:常见的移植途径包括结肠镜、鼻肠管、口服胶囊等,各有优缺点;粪菌菌液的制备、冻干保存等剂型标准化问题尚未完全解决,影响疗效稳定性。③ 作用机制不明确:FMT 是复杂的“多微生物移植”,其疗效究竟是特定菌株、菌群整体还是代谢产物介导的,尚未完全阐明,难以实现精准化治疗。④ 安全性与长期风险:尽管总体安全性较好,但仍有报道 FMT 后发生严重感染(如肠出血性大肠杆菌感染)、自身免疫疾病等不良反应,其长期影响仍需长期随访研究。⑤ 适应症拓展与疗效评估:在 IBD、肠易激综合征、代谢性疾病等复杂疾病中的疗效尚存在争议,需要更多高质量临床试验证据,并建立统一的疗效评价标准。